この記事は今年読んだ一番好きな論文 Advent Calendar 2015の23日目の記事です.
今日紹介するのは,Journal of Chemical Information and Modelingという論文誌に掲載されたAn Inexpensive Method for Selecting Receptor Structures for Virtual Screeningという論文です.日本語で言うと,「バーチャルスクリーニングで使うタンパク質の構造を割と軽めの計算で選ぶ方法」というものです.
Huang Z, Wong CF. J Chem Inf Model. (in press), doi:10.1021/acs.jcim.5b00299
Publication Date (Web): December 14, 2015
http://pubs.acs.org/doi/10.1021/acs.jcim.5b00299
先日アクセプトされたばかりでまだ著者原稿版しか載っていませんが,僕らがやっている研究に近いというか,なんで思い付いてさっさと投稿しなかったんだというツッコミを自分に入れながら読んでいました.
1 バーチャルスクリーニングとは?
バーチャルスクリーニング (virtual screening) とは創薬分野で主に使われる単語で,計算機で薬の候補になりそうな化合物を選別 (screening) することを指します.薬の候補になりそうかどうかは,あるターゲットのタンパク質に対して活性が有るか無いか,という指標で図られます.この活性の有無を予測して選別することが目的です.
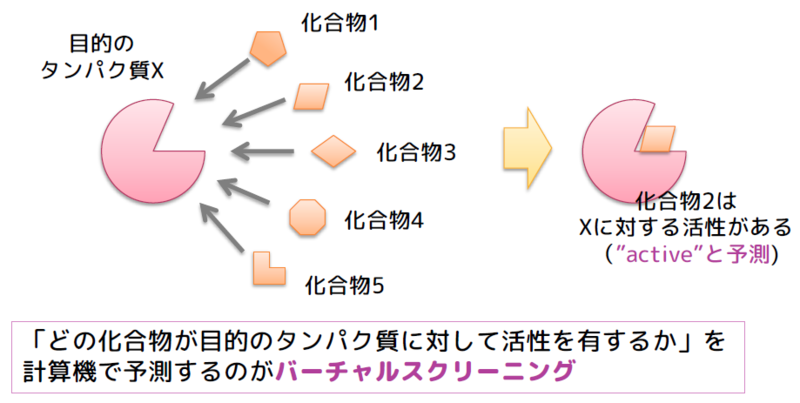
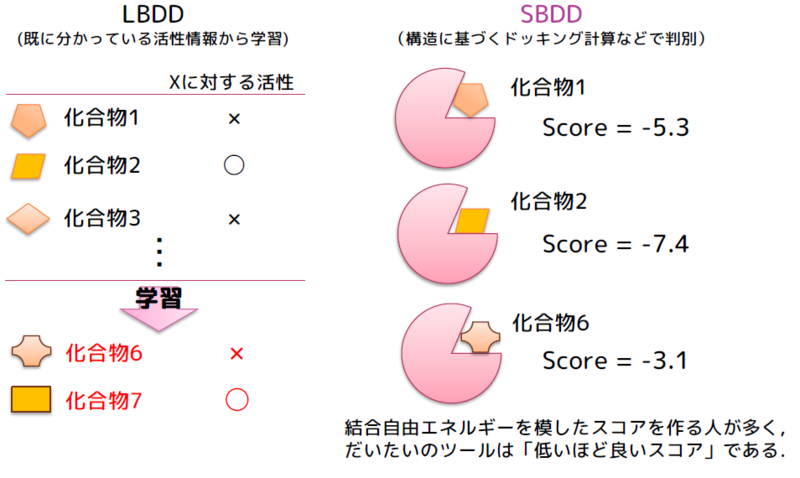
バーチャルスクリーニングには大きく2つの方法があり,化合物の形と既に分かっている活性の情報(教師情報)から未知の化合物に対する活性を予測するligand-based drug design (LBDD) と,ターゲットとなるタンパク質の立体構造情報を使ってドッキングシミュレーションなどの物理化学的な計算を用いるstructure-based drug design (SBDD) があります.それぞれ一長一短ありますが,今回紹介する論文は後者のSBDDのお話です.
2 この論文はどういう問題を扱ってるの?
SBDDでは,ターゲットとするタンパク質の立体構造がとっても重要です.立体構造を決めた人はProtein Data Bank (PDB) というデータベースに登録していくのですが,同じタンパク質でもいろんな立体構造があるので,それらは個別にそれぞれ登録されています.構造屋さんはリゾチームが大好きなので,例えばリゾチームを見てみると700個くらい立体構造がPDBに登録されています(参考:http://d.hatena.ne.jp/tonets/20120730/1343655777).
そのため,「あるタンパク質Xを阻害する化合物を探したい!」と言っても,タンパク質Xの構造データはたくさんあるので,そのうちどれを使ってSBDDすればいいのか分かりません.化合物がはまりそうなポケットに何か既にはまっているもの(ホロ体といいます)だと,そうでないもの(アポ体)よりも良さそうですが,一概には言えません.どの構造がバーチャルスクリーニングに適しているかを選ばなければなりません.

上の図はイメージ図ですが,実際の構造もちょっとずつ違っています.CDK2タンパク質を例に見てみましょう.

ほとんど同じと思う人も多いかと思いますが,ちょっとずつ違っています.
3 1番単純な方法
さて,構造の選び方ですが,1番シンプルな方法はこんな感じです.
タンパク質Aについて既に活性がある化合物(active)と,活性がない化合物(inactive)または活性が多分ない化合物(decoy)を集めてきて,実際にタンパク質Aの構造A1, A2, ...とドッキングさせて「activeの評価が高く,inactive/decoyの評価が低くなる」ようなタンパク質の構造を選べば良い.
図にするとこんな感じです.化合物がスコアの良い順に並んでいると思って下さい.
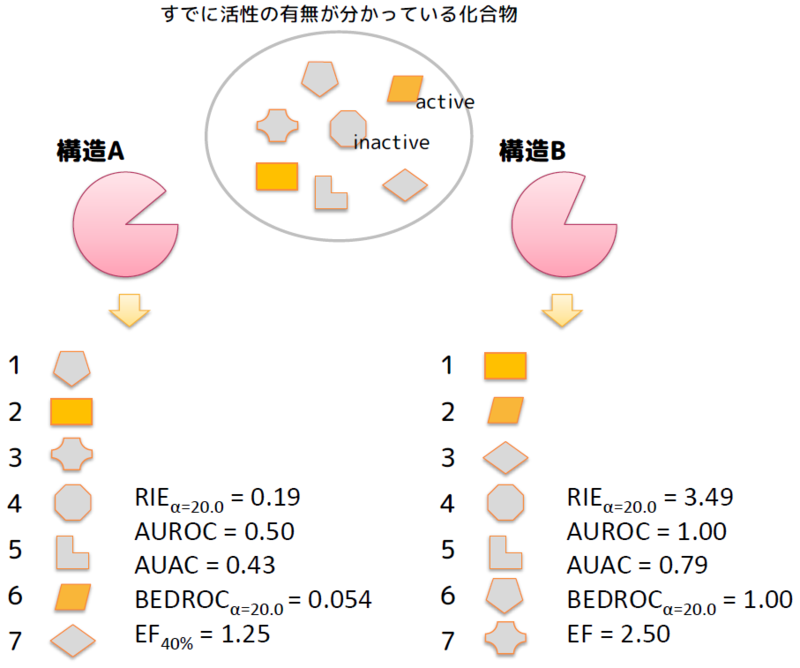
図中にでているRIE,AUROC,AUAC,BEDROC,EFはどれもランキングの良さを表す指標で,上位にactiveが来れば来るほど大きな値になります.詳細はhttp://d.hatena.ne.jp/tonets/20140604/1401856579とか見て下さい.
いろいろ指標を出しましたが,まぁ人の目で見ても「構造B」が良さそうというのが分かりますね.
4 1番単純な方法の問題点
この方法は1番単純でかつ確実な方法なのですが,計算が大変という問題もあります.図ではactiveが2個でinactive (decoy) が5個ですが,実際にはactiveが数十個,inactive (decoy) は数千個というレベルで計算させることが多いです.単純に数が多いので大変,ということですね.
5 この論文が提案したこと
この論文では,「activeとinactiveを全部ドッキングするのは大変だから,activeだけの結果からなんとか判断しよう」としました.5つの指標を提案していて,そのうちScreening Performance Indexと名付けた5番目の指標が1番良かったと言っています.SPIの式をそのまま引用します.
はactiveの数,
はドッキングがちゃんとできたactiveの数ですがほとんど
と同じです.
は
を全ての候補の構造で足し合わせたもので,構造が
個あって
個のactive化合物が全てドッキングできたとすると
となります.
式で見るとちょっと複雑そうですが,要するにactiveだけドッキングした結果の全体平均スコアよりも高いスコアになったactiveをたくさん得た構造が勝ち,ということです.図にするとこんな感じです.

この図では,平均が-8.7で,構造Aは長方形の化合物だけ,構造Bは2つとも平均より良いスコアなので,構造Bの方がSBDDに適していると言うことができます.
6 SPI (Screening Performance Index) を使った結果
さて,本当にこのSPIという値でバーチャルスクリーニングに適した構造を選ぶことができるのでしょうか.詳細は割愛しますが,この論文では8種類のタンパク質に対して,それぞれ10〜30個くらいの構造を用意して,activeだけを使ってSPI値で選んだ構造が,実際にactiveとinactiveの両方を使って計算したBEDROC/RIE/AUAC/1%EF/10%EFとどのくらい相関するかを示しています.結果的にはSPIとBEDROCが平均して0.87ほどの相関係数を持つことが示されました.つまり,activeだけで選んだ構造は,実際にactiveとinactiveの両方で検証しても識別能が高かったということになります.
興味深いのは,結合部位の体積やタンパク質構造の解像度,アポ体/ホロ体の区別とはあまり関係がなさそうだったということです.このあたりは特徴付けが難しいのですが,構造の特徴そのものから識別能が分かるようになると,activeとのドッキングすら要らなくなるので,More Inexpensiveな方法で構造を選ぶことができるようになります.この論文の将来展望といったところでしょうか.
7 あとがき
細かい方法論の論文を紹介してしまいましたが,意外に誰もやっていなかった話(もしくはみんな暗黙のうちにやっていたかもしれない方法)をうまく論文化したなぁという印象です.ちなみにこのJournal of Chemical Information and Modelingという論文誌は,JACSで有名なACSが刊行する雑誌で,ケモインフォマティクスを中心に,分子シミュレーションやバイオインフォマティクスの方法論も数多く載せています.バーチャルスクリーニングとか言い出す人はまず読んでいる雑誌なので,もしこういった分野に興味がありましたら論文を眺めてみると良いかもしれません.