ランク学習でバーチャルスクリーニングする
この記事は創薬 (dry) Advent Calendar 2019の20日目の記事です。
ランク学習という機械学習手法でバーチャルスクリーニングする話をします。
ランク学習とは?
ランク学習とは、順序関係を学習する機械学習です。
機械学習では、0か1を学習する「分類」と、目標変数の値を学習する「回帰」が有名ですが、その中間のような存在です。

ランク学習は順序=ランキングを出力するので、0か1の分類よりは詳しい情報が得られます。一方で、具体的に「IC50が◯nM」といった値までは予測できません。そういう意味で、分類と回帰の中間のような存在なわけです。
ランク学習の詳細については、こちらを見るとだいたいわかります。sz_dr先生によるつよいAdvent Calendarです。

バーチャルスクリーニングで何ができればよいのか?

バーチャルスクリーニングは、化合物の活性や物性などが良さそうなものを、膨大な化合物ライブラリの中から選抜する作業です。数千万化合物とかの中から、実際に実験する価値がありそうな数百とか数千とかの化合物を、計算機(と職人芸)で選びます。
実際に実験に回せる化合物の数には限度があります。実験の種類や予算にもよりますが、とりあえず1000個検討するみたいなことが多いです。ライブラリが1000万化合物だとすると、1000化合物は0.01%。「It's like looking for a needle in a haystack. (まるで干し草の山の中から縫い針を探すようだ)」と言われる所以でもあります。
一方で、似たような状況が、情報検索と呼ばれる分野で一足先に研究されていました。膨大なウェブページの山から、検索窓の単語にマッチするウェブページを検索するGoogleやYahoo!などの検索エンジンです。ユーザーが期待するウェブページを瞬時に上位に示す検索エンジンの技術は、化合物のバーチャルスクリーニングにも応用できる、できそう、な気がしませんか?
つまり、こういうことです。

どうやってランク学習するのか?
ランク学習にもいろんな手法があります。ここでは2値分類でよく使われるSVMのランク学習版であるRankSVMというのを紹介します。詳細はsz_dr先生の記事で充分なのですが、簡単に説明しておきます。

SVMでは、判別平面からのサンプルの距離である「マージン」を最大化する最適化をしています。
RankSVMでも、似たような距離の最大化をしています。ランク学習ではサンプル間の順序関係がわかっているので、その順序を満たすように1列に並べるようなベクトルを探します。
図では、 という順序関係があるので、この順番に1列に並べられるベクトルを探します。(
は順序関係を表す記号です。数値であれば不等号
、集合の包含関係であれば
に相当するような関係を表す記号です。)
1列に並べるベクトルはいくつもできますが、「できるだけサンプル間の距離が離れるように」並べます。正確には、一番近いサンプル間の距離ができるだけ大きくなるように並べ方を最適化します。
図では、の並べ方では
と
の間が一番近くなっており、
の並べ方では
と
の間が一番近くなっています。この2つを比べると、
の並べ方での
と
の間の距離の方が大きいので、
よりも
の方が良い、というように学習します。
 これでランク学習を使って、化合物のスクリーニングをすることができるようになりました。ばんざい。
これでランク学習を使って、化合物のスクリーニングをすることができるようになりました。ばんざい。
無意味っぽい順序関係を無視する
ところで、化合物のスクリーニングでは、順序関係をそのまま使わない方が良いことがあります。
具体的には、こういう状況を想定しています。(パーセンテージの数値はたとえば10 uMでの阻害活性測定実験とかを想像してください)
 この、「活性が無い2化合物の順序関係」と「活性値がめっちゃ似てる2化合物の順序関係」は、学習するときに使わないでおこうという作戦です。
この、「活性が無い2化合物の順序関係」と「活性値がめっちゃ似てる2化合物の順序関係」は、学習するときに使わないでおこうという作戦です。
実装は、確率的勾配降下法を使っての最適化を逐次更新しながらやる方法で実装できます。

具体的なコードは以下にあります。 szdr.hatenablog.com
どのくらい性能があがるのか?
PubChemのInhibition assayのセットを5つ持ってきて、普通にRankSVMするのと、無意味っぽい順序関係を無視してRankSVMするのを比べてみました。データセットはこちらです。

評価指標はNDCG1@100、評価方法は5-fold Cross Validationです。NDCG1@100の定義は以下にあります。
szdr.hatenablog.com
結果は以下のようになりました。
 表の見方は、cutoffのon/offが似た活性値の化合物間の順序を無視する(on)かしない(off)か、ignore negativeのon/offが活性無し化合物間の順序を無視する(on)かしない(off)かに対応します。
どのデータセットでも、2つの無意味っぽい順序関係を無視して学習したほうが良い予測ができるという結果になりました。
表の見方は、cutoffのon/offが似た活性値の化合物間の順序を無視する(on)かしない(off)か、ignore negativeのon/offが活性無し化合物間の順序を無視する(on)かしない(off)かに対応します。
どのデータセットでも、2つの無意味っぽい順序関係を無視して学習したほうが良い予測ができるという結果になりました。
おわりに
ランク学習は、情報検索分野を超えて、バイオインフォマティクス分野やケモインフォマティクス分野での応用が進んできています。個人的には、ランク学習と相性の良い問題がバイオ・ケモ分野にゴロゴロ転がっていると思っているので、興味を持ってもらえると嬉しいです。

おまけ
本記事は、以下の論文の内容の紹介でした。もう少し詳しい内容が以下に書かれています。 www.sciencedirect.com
Masahito Ohue, Shogo D. Suzuki, Yutaka Akiyama: Learning-to-rank technique based on ignoring meaningless ranking orders between compounds, Journal of Molecular Graphics and Modelling, 92:192-200, 2019. doi: 10.1016/j.jmgm.2019.07.009
おまけ2
szdr先生のブログ便利すぎワロタ
更新履歴
2019/12/14 目線が不適切だったので消しました
IQ1の助教
この記事はIQ1 Advent Calendar 2019の4日目の記事(遅刻してごめんなさい)です。
助教とは
「助教」とは、大学の教職員の1職名です。
東京工業大学においては、研究・教育に携わる職員には以下のような職位があります。
教授 Professor
准教授 Associate Professor
講師 Associate Professor (Lecturer)
助教 Assistant Professor
栄誉教授 Honorary Professor
名誉教授 Professor Emeritus
特命教授 Institute Professor
卓越教授 Distinguished Professor
特任教授 Specially Appointed Professor(※)
特任准教授 Specially Appointed Associate Professor
特任講師 Specially Appointed Associate Professor (Lecturer)
特任助教 Specially Appointed Assistant Professor
リサーチ・アドミニストレーター University Research Administrator
特任専門員 Specially Appointed Expert
研究員 Research Staff
技術限定職員 Specifically Assigned Technical Staff
事務限定職員 Specifically Assigned Administrative Staff
教務支援員 Student Affairs Support Staff
技術支援員 Technical Support Staff
事務支援員 Administrative Support Staff
特定教授 Visiting Professor
特定准教授 Visiting Associate Professor
特定講師 Visiting Associate Professor (Lecturer)
特定助教 Visiting Assistant Professor
客員研究員 Visiting Scholar
非常勤講師 Part-Time Lecturer
IQが1なので違いは詳しい違いは説明できません。しかたないね。
一昔前はよく似た「助手」という職位がありましたが、今はありません。
「助教授」と間違えられますが、「助教」です。「助教授」も今は存在しておらず、相当するのは「准教授」です。
今回のIQ1 Advent Calendar 2019には助教が4人ほど混じっているように見えます。
IQが1の助教がたくさんいるということで、大変頼もしいですね。
助教のお仕事とは?
助教は何をする人なのか。
我々はその謎を解き明かすべくジャングル(大岡山)の奥地へと向かった。。。
レポーター「こんにちは!⚫⚫先生はいらっしゃいますか?」
???「あ~、今日は来ていないみたいですね」
。。。
助教のお仕事とは?(2)
助教というお仕事は、研究や教育をすることです。
毎日研究したり授業したり、研究室にいる学生に指導をしたり、議論をしたり、学会に行って調査したり成果を発表したり、世界中の研究者と一緒に研究したりします。大学内の設備の管理をしたり、新しい教育カリキュラムを考えたりもします。人によっては研究室の主宰者(Principal Investigator, PI)として研究室運営もやります。本を書いて出版する人もします。
研究にはお金が必要なので、この研究めっちゃ大事なんですお金くださいと色んなところに頼み込みに行ったりもします。研究の大事さを語るためにプレゼンや資料をたくさん準備したりします。
1個人の裁量に委ねられている面が強いので、助教と言ってもやっていることは人それぞれです。詳しくは助教と呼ばれている人に聞いてみましょう。
tonetsのお給料
まああんまり変わっていませんが、2019年の収入もほぼ確定しそうです。
もうめんどくさいので11月時点のをオープンにします。私は2015年4月に初めて教員として着任した、現在助教5年目の、32歳、男性です。2014年の1年間の研究職経験(ただし学振PD=雇用関係無し)がありました。

いくつか補足が必要な点を挙げます。
- 裁量労働制という制度なので、定時や残業という概念はありません。
- 基本給月額は、級と号俸によって決まります。級は助教であれば2級確定で動きません。号俸は年1ある評価で、S/A/B/C/Dのうち「C(標準)より上」を貰えれば上がります。(CおよびDはいわゆる昇給がありません)
- 年俸制(旧)という制度なので、ボーナスという概念が、月々支払われる業績給というものに吸収されています。したがって見た目のボーナスはありませんし、定額が年に12回支払われます。業績給は年1ある評価によって大きく変動します。
- 年俸制(旧)という制度なので、退職したときに払われる退職金が、業績給の中に含まれます。したがって、私は退職時にもらえる退職金はありません。
- 任期付の教員なので、任期付教員特別手当を頂いています。任期付教員特別手当は通算で5年目までもらえます。それ以降は任期付でも貰えません。助教は20,000円です。(教授10,000円、准教授15,000円、講師15,000円)
- 4歳の子どもを1人扶養しているので扶養手当を頂いています。10,000円です。妻はガッツリ働いているので扶養手当の対象ではありません。
- 東工大では、教育コースを担当する教員や、博士後期課程学生の主指導教員に、大学院調整費というものが支給されます。主指導学生の数と、職位によって変動します。私は博士後期課程の主指導学生は0人ですが、教育コースを担当しているので最低額が支給されています。
- 都市手当というものが存在します。(基本給+大学院調整額+高校教員調整額+管理職手当+扶養手当)✕18.8%で計算されます。
(322,300+10,500+0+0+10,000)✕0.188 = 64,446.4(切り捨て)となります。 - 控除項目としてなんかいっぱい引かれます(IQが1)
- 給与支給累計がいわゆる年収になります。ここには通勤手当は含みません。
- 12月の支給合計額も、669,904円からほぼ変わらないはずです。通勤手当が3,943円なので、いわゆる2019年の年収は、7,243,478+669,904-3,943=7,909,439円(概算)となるかと思います。
おわりに
いかがでしたでしょうか?
研究者の仕事の様子はtwitterを見ればわかりますが(ほんとう?)、だいたいこんな感じで生きています。
助教の年収は400~600万円!仕事内容やなる方法について解説
https://career-picks.com/average-salary/jokyo-nenshu/
という記事や、
という記事がありますので、退職金を含むとはいえ、ありがたいことに助教の中では恵まれている方かもしれません。
IQが1なりにこれからも頑張ります。現任期の残りはあと4ヶ月です。
4年間働いた東工大を退職しません
退職しませんエントリです。
題名のままです。
しかし、私の任期は5年間なので、2019年度中になんらかのアクションをしなければなりません。
一体私、これからどうなっちゃうの〜!
2018年の収入
助教3年目の12月ですどうぞ。 pic.twitter.com/4qSOwU4pca
— 猫じゃら美少女 (@tonets) December 18, 2017
2017年の年収はこれくらいでした。
2018年の年収は、

でした。来年はもう少し上がると良いな。
追記:忘れてたけど、これの+数%分くらいの、兼業収入(単発の講演の類、印税等)があります。
IQ1の結婚記念日
この記事は IQ1の2まいめっ Advent Calendar 2018の20日目の記事です。
本日12月20日は私tonetsの結婚記念日です。
結婚してから5年が経ちました。つまり、2013年12月20日に結婚したわけです。
2013年12月20日といえば、tonetsはD3で、D論発表(公聴会)の5日前でした。
その日は区役所に行ったこと以外はあまり覚えていませんが、前日と翌日にそれぞれTSUBAME 2.5を420ノード予約していることを示すGoogle Calendarのメモは残っていました。
IQが1しかなくても、相手がいれば結婚できます。
結婚に必要なものは、ぐぐれば調べられます。
こちらのサイトのコピペですが、
- 婚姻届 1通
- 戸籍謄(抄)本 (もとの本籍地でない役所に届ける場合)
- ふたりの旧姓印鑑
- ふたりの身分証明書
- 20歳以上の証人ふたりの署名と捺印
が揃っていれば、婚姻届をAcceptしてくれます。不備があったらMinor Revisionです。
婚姻届のテンプレートは、任意の役所の戸籍担当のところで「ください」と言えばくれます。
証人も任意です。大学院生の友人とか、そのへんのおっさんでも構いませんが、実用上は親とかが選ばれることが多いです。
役所の人は窓口にIQ1の人が現れても対処できるように訓練されてますので、IQが1しかなくても、大学院生でも、誰でも結婚できます。相手さえいれば。
結婚に関する一般的な知識をサーベイしたい場合は、結婚の専門誌である「ゼクシィ」を読めば大丈夫です。CellとかNature Chemistryみたいなもんです。
結婚CFPの解説でした。
おしまい。
IQ1のD進リターンズ
この記事はIQ1 AdC 2018の19日目の記事です。
参考記事
みなさん。
IQが1なので僕はD進しました。
なぜD進するのでしょう?
就活が面倒だったからです。
研究が楽しかったからです。
IQ1にリクなんとかとかマイなんとかはレベルが高すぎます。
D進しない人はなぜD進しないのでしょう。
D進しない人は、早く働きたいと言います。
しかしD進しない人は、だいたい修士=M進はします。
Mは研究することが課せられます。Dと同じです。
M進するくらいならD進すればいいのにと思います。
働きたいなら学部を出てすぐに働いた方が、生涯のお金はたくさんもらえます。
年収ならD卒が1番高いです。
いまやあたりまえのようにD卒者がいます。
D卒じゃないと相手にされない企業もあります。
人はなぜM卒にこだわるのか。
IQが1なのでわかりません。
おしまい
タンパク質間相互作用予測の話

この記事は創薬 Advent Calendar 2017 (http://adventar.org/calendars/2412) の14日目の記事です。
連載3日目は、化合物を選ぶバーチャルスクリーニングから少し離れて、タンパク質間相互作用という生命現象を予測する話をします。
タンパク質間相互作用と創薬
タンパク質間相互作用、Protein-Protein Interaction、略してPPIとよく呼ばれます。狭義にはタンパク質同士が結合して複合体を形成すること、広義にはタンパク質同士がなんらかの影響を及ぼし合っていることを指します。創薬としては、PPIするタンパク質同士をPPIさせなくするような薬=PPI阻害薬を開発できないかと、ここ10年以上[要出典]、試行錯誤がされています。
話題の抗体医薬品であるオプジーボ(ニボルマブ)も、PD-L1とPD-1というタンパク質同士のPPIを阻害するので、PPI阻害薬と呼ぶことができます。が、一般に抗体医薬のことをわざわざPPI阻害薬と呼ぶことはほとんどありません[要出典]。抗体医薬が狙うのは細胞膜上に刺さっている膜タンパク質(オプジーボであればでT細胞のPD-1)であり、この膜タンパク質に覆いかぶさってはたらきを阻害します。しかし、2000年代から注目されている[要出典]PPI阻害薬の創薬は、細胞の中のPPIを標的として、細胞膜を通過できる低分子などで薬を作ろうというものですので、抗体医薬の創薬とは少し毛色が異なってきます。
なお、抗体医薬については中外製薬のウェブサイトがわかりやすいです。(たとえばこちら https://chugai-pharm.info/bio/antibody/antibodyp14.html)
狭義のPPI、タンパク質の複合体には、いろいろな形が存在します。構造ベース創薬をやるなら複合体構造の形を知りたくなりますが、すでにたくさんの複合体構造が解かれてProtein Data Bankに登録されています。以下はその一例です。(Scott DE, et al. Nat Rev Drug Discov 15: 533, 2016 より引用)

この中で、たとえばインターロイキン2 (IL-2) については低分子の阻害剤の設計がいくつか論文等でも報告されています。(Scott DE, et al. Nat Rev Drug Discov 15: 533, 2016 より、一部改変)
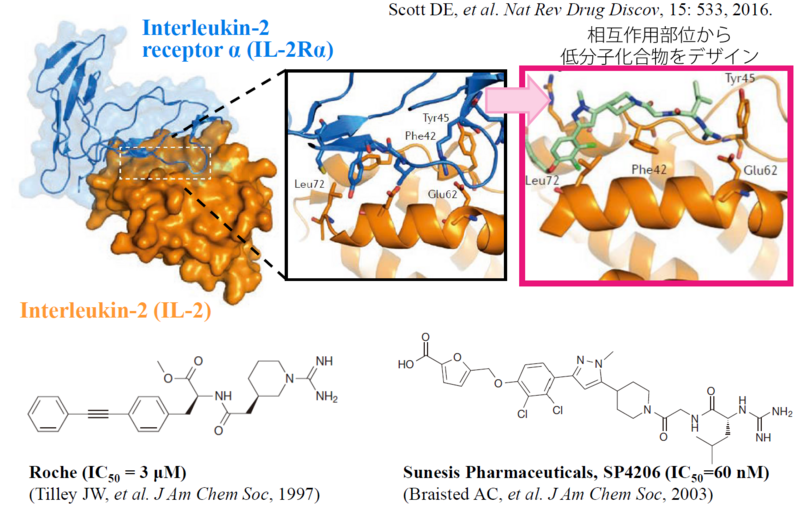
細胞膜上のタンパク質は抗体医薬によって数多く狙われていますが、細胞の中のタンパク質にはまだまだ手付かずのものも多いです。PPI阻害薬は、創薬過程にたくさんの課題はありますが、新奇の標的が狙えるとして注目されているのです。
タンパク質間相互作用を予測する
タンパク質間相互作用の予測は、化合物の世界のバーチャルスクリーニングと様相がよく似ています。すでにPPIの相方が分かっているタンパク質同士を機械学習にぶち込んで未知のPPIを予測する機械学習ベースの方法と、ドッキングなどの方法で立体構造から相方を探す構造ベースの方法が存在します。
機械学習ベースの方法は、創薬 Advent Calendar 2017 (http://adventar.org/calendars/2412) 13日目の記事として紹介した薬剤標的相互作用予測 (http://d.hatena.ne.jp/tonets/20171213/1513094866) の方法がほぼそのまま使えます。つまり、タンパク質Aとタンパク質Bのペア (A, B) をなんらかの方法で特徴ベクトルにし、Y2Hなどの実験によって得られた相互作用の有る無しを {+1, -1} としてラベル付けし、SVMや無敵のディープラーニング[要出典]に突っ込んであげればよいのです。
実際に2006〜2007年ごろから機械学習によるPPI予測が試みられて、おそらく現在までに50報くらいは論文があると思います。昔は"-1"のラベル、「相互作用しないよ」というラベル情報がほとんどなく、相互作用するペアの相方をランダムに入れ替えたものを便宜的に使っていたりしましたが、最近では「相互作用しない」という情報を集めたデータベースなんかもでてきています(http://mips.helmholtz-muenchen.de/proj/ppi/negatome/)。
構造ベースの方法は、タンパク質同士の複合体構造を予測し、そのときのエネルギースコアの値を使って相互作用するかしないかを判定します。タンパク質同士の複合体を予測すると言った論文もこれまた山ほどあります(ざっと数百報の規模)。実は計算機による複合体予測の歴史はずっと古く、はじまりは1972年と言われています。以下の図は、私が調べた中で世界初だと思う複合体予測(BPTIとα-chymotrypsin)の論文 (Blow DM, et al. J Mol Biol 69, 137-144, 1972) の図です。

今ではたくさんのフリーソフトウェアやサーバーが出てきて、とても気軽に複合体構造が予測できるようになりました。例えばClusPro (http://cluspro.bu.edu/) は、タンパク質複合体構造予測コンペのCAPRIでとても優秀な成績を収めているサーバーで、よく使われています。Glideで有名なSchrödinger社がPIPERという名前で販売しているものと中身はほぼ同じです。
1つのタンパク質ペアに着目して複合体構造を予測したり、数ペア程度のタンパク質のPPI有る無しを知りたいという場合には、ClusProを使うのが良いかもしれません。しかし、例えば調べたいタンパク質のペアが1,000とか10,000とか、百万通りとかある場合には、なかなか構造ベースの方法では計算が大変で追いつきません。
MEGADOCK

そんな要望に応える形で我々が開発したのがMEGADOCK (http://www.bi.cs.titech.ac.jp/megadock/) です。MEGADOCKは、ClusProほどの精度は無いものの、速くたくさんのタンパク質ペアのドッキングが計算ができる唯一のソフトウェアです。流行りのGPU計算やスパコンでの計算にも対応しており、たとえば1台に4基のGPUが刺さったワークステーションでもGPUをすべて使って計算することができます。
しかし、ご家庭にスパコンはありません。自分の知りたいタンパク質ペアがどういう予測結果になるのか、かんたんには計算ができない場合もあるでしょう。そんなときのために、MEGADOCKの計算を予めやっておいたデータベースも作っています (MEGADOCK-Web: http://www.bi.cs.titech.ac.jp/megadock-web/)。論文はopen access誌のBMC Bioinformaticsという論文誌に2018年にpublishされることが決まっておりますので、興味のある人はpublishされたら読んでみてください。中身としては、ヒトのタンパク質鎖約7500個の全対全、ペアのPPI予測結果と複合体モデル構造が閲覧できるようになっており、JavaとPlay Frameworkを用いて構築されています。

まとめ
最後は脱線気味でしたが、新しいPPI標的を探して、PPI阻害薬を設計していくのは、創薬としてはかなり壮大な旅です。そんな旅を支えるツールとして、様々なソフトウェアが開発され、利用されていますので、興味がわきましたら是非調べてみてくださいね。