エスプレッソ(Spresso)の話

この記事は創薬 Advent Calendar 2017 (http://adventar.org/calendars/2412) の12日目の記事です。
Spressoという計算ツールの紹介です。論文はこちら。
Yanagisawa K, Komine S, Suzuki SD, Ohue M, Ishida T, Akiyama Y. Spresso: An ultrafast compound pre-screening method based on compound decomposition, Bioinformatics, 33(23): 3836-3843, 2017.
https://academic.oup.com/bioinformatics/article-lookup/doi/10.1093/bioinformatics/btx178
(オープンアクセスです)
ツールのURLはこちら : http://www.bi.cs.titech.ac.jp/spresso/
はじめに
創薬の初期段階に、計算で有望な化合物を選び出すという過程(=バーチャルスクリーニング)があります。
バーチャルスクリーニングにはいろんなアプローチがあり、毎日のように新しい手法やアルゴリズムの論文が出版されています。
Spressoも、そんなバーチャルスクリーニングのためのツールの1つです。
Spressoが前提とするのは、
- 分子標的薬の設計である
- 標的となるタンパク質の構造が解かれている
- 化合物ライブラリがある
の3点です。いわゆる「構造ベースのバーチャルスクリーニング(structure-based virtual screening, SBVS)」をやります。何も無いところから新しい化合物を生み出す最近流行りのde novo設計はできませんが、たくさんの(数千万個の)候補化合物の中から、構造ベースで、高速に、有望な化合物を選ぶことができます。
しくみ
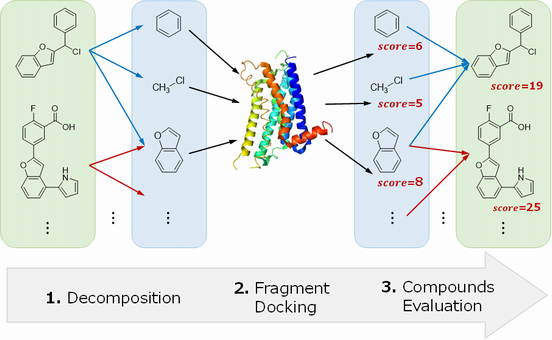
Spressoのしくみを説明します。
- まず化合物を、フラグメントにブツ切りにします。
- 次に、ブツ切りにしたフラグメントとタンパク質を、ドッキングツールでドッキングします。ドッキングツールはGlideでもGoldでもAutoDockでもmyPresto/sievgeneでもなんでも良いですが、Spressoが現行でサポートしているのはGlideのみです。
- 最後に、フラグメントのドッキングスコア(GlideScoreなど)を使って、ある数式に基いて元の化合物の有望度を計算します。
以上です。図にするとこんな感じです。
なぜフラグメント?
色々と疑問が出てくると思いますが、まずは「わざわざフラグメントにブツ切りにする」ことのメリットから説明しましょう。
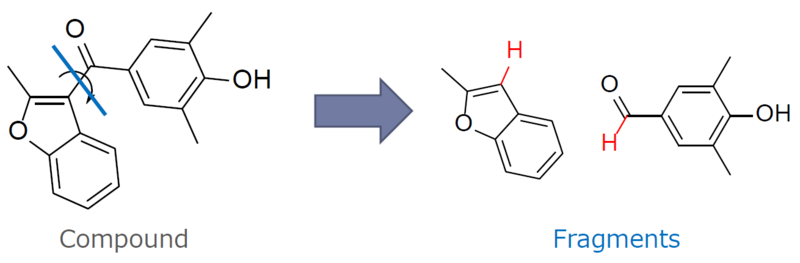
世の中に化合物はたくさんあるのですが、単純にすべてをドッキングしようとすると結構大変です。たとえばZINCと呼ばれる有名な化合物ライブラリには、化合物がだいたい2千万個とかあります。ドッキング自体は1化合物あたり1秒くらいで終わりますが、2千万個の化合物だと5500時間=230日ぐらいかかる計算です。もちろん、230台の計算機があれば1日で、460台の計算機があれば半日で、920台あればたった6時間で終わりますが、なかなか大変です。しかし、化合物を構成するフラグメントの種類は、化合物に比べるとそんなに多くないのです。

たとえばこの例では、28,629,602化合物が263,319種類のフラグメントだけで構成されていることがわかりました。要するに、フラグメントのなんらかの計算結果を使って化合物を評価するならば、263,319種類のフラグメントの計算だけをやっておき、あとは計算結果を再利用すればよいということになります。
フラグメントから化合物にどうやって戻す?
さて、次の疑問は「どうやってフラグメントに切ったものから化合物に戻すのか?」ですかね。
Spressoでは、フラグメントから化合物の形に戻すことをしません。なので、有望な化合物を選ぶだけでなく、結合構造を知りたい人はSpressoは(ほぼ)使えません。
フラグメントのドッキングスコアから化合物の評価をするには、たとえばフラグメントのドッキングスコアを単純に足したり、最良値をとったり、平均値をとったりすれば良いことになります。色々試した結果、3乗一般化総和(フラグメントスコアを3乗した値をすべて足して3乗根を取る)が一番良かったのでそれを使っています。
ここで、はフラグメントを、
はフラグメントのドッキングスコアを表します。
はある化合物に着目したときに構成されているフラグメントのすべてについて総和を取る操作を表します。
Spressoの性能
最後に、肝心の結果についてです。
まず計算速度ですが、ZINCの28,629,602個の化合物をGlide HTVSモード(速いモード)でドッキングした場合、およびSpresso中でGlide SPモード(普通モード)を使った場合のSpresso、Spresso中でGlide HTVSモードを使った場合のSpressoを比較したグラフが下の図です。

ドッキングの回数が減るので当たり前なのですが、SpressoはGlide HTVSに比べて100倍前後速いです。
次に精度です。精度はDUD-Eというバーチャルスクリーニング用のデータセットから102セットをすべて使いました。少しややこしいのですが、Glide HTVSで全体のをスクリーニングしてからその後にGlide SPで上位1%を取った場合、Spresso(SP利用)で全体の
をスクリーニングしてからその後にGlide SPで上位1%を取った場合、Spresso(HTVS利用)で全体の
をスクリーニングしてからその後にGlide SPで上位1%を取った場合、のEnrichment Factor (EF1%) を調べました。これはSpressoやGlide HTVSが、プレスクリーニングツールとして利用されることを想定しているためです。
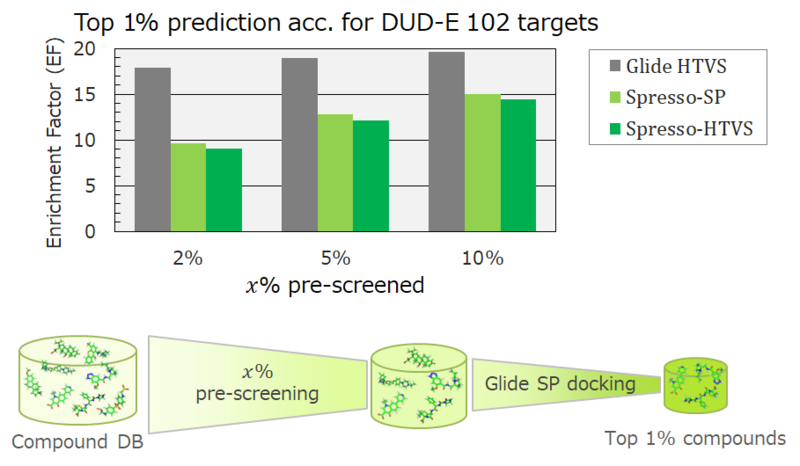
これを見ると、SpressoはHTVSの半分くらいの精度であると言えます。の数字が10%くらいになると、HTVSに近づいていきますが、その後のGlide SPモードによるスクリーニングに時間がかかってしまうので、お手軽さは減ってしまいます。
まとめ
Spressoは、精度はGlide HTVSの5〜8割くらい、速度はGlide HTVSの100〜200倍くらい、と覚えて貰えれば良いと思います。構造ベースの大規模スクリーニングがお手軽に、ご家庭でできるレベルになってくれれば、という思いが名前に込められています。
謝辞:この記事の図のほとんどは @yanagi3150 https://twitter.com/yanagi3150 によるものです。
小咄
- 本成果はGIW2016という査読付き国際会議で最初に発表されました。発表当初はESPRESSOと名乗っていましたが、同じ名前のツールが存在していたため、名前が変わりました。
- GIW2016に提出した論文原稿は、査読の結果採択されました。その後、論文誌推薦としてBioinformatics誌に推薦され、Bioinformatics誌の査読を受け直しました。major revisionでかなり大変なリバイズをしましたが、@yanagi3150 の頑張りで無事acceptに漕ぎ着けられました。
- 実は2017年3月30日に論文が公開されていましたが、Bioinformatics誌のGIW2016 Special Issueに載せる予定の論文が揃うのに時間がかかり、結局正式なpublishは2017年12月1日となりました。GIW2016があったのが2016年10月なので、1年越しの刊行です。