IQが1のD進
この記事はIQが1 Advent Calendar 2017 (http://adventar.org/calendars/2377) の13日目の記事です。
D進=大学院博士後期課程への進学について書きます。
だいたい理工系(特に情報系)を想定してます。医歯薬系・文系はよく知りません。
・D=博士後期課程ってなに?
▶︎修士課程の後にさらに3年間ほどあるやつです。
・大学の後の修士の後にまだ何すんの?
▶︎研究をしたり、専門分野の教育を受けます。世界の最先端の研究を行い、まだ誰も知らない未知の発見を学術誌の論文や博士論文=博論=D論という形で世界中の人々に教えてあげることを目指します。
・どうしたらD進できるの?
▶︎願書を出して入試を受けてください
・どうしたら卒業(修了)できるの?
▶︎博士後期課程は、学位=博士=Dを取得することが目的です。Dが取れたら修了です。Dが取れないけど講義の単位だけ揃えて課程を辞めることを単位取得満期退学とか言います。戦略的にこのルートを選ぶ人もいますが、割愛します。
・どうしたらDが取れるの?
▶︎いくつかの敵を倒すとDが取れます。
・敵とはなんですか?
▶︎大学・学科(部局)等で決まりはバラバラですが、ほとんどの学科では学術雑誌に論文を1本以上掲載する必要があります。どんな雑誌でも良いわけではなく、査読があり、英文誌であることが大前提です。ただし、国際会議のプロシーディングでも良い場合もありますし、レター論文という3〜4ページくらいの短い論文は認められないこともあります。3本くらい求められる学科もあるようです。
・査読とはなんですか?
▶︎誰かがあなたの論文を読んで、おかしいところがないか確認します。
・ほかに敵はいますか?
▶︎審査があります。卒論発表の長い版とでも思ってください。一般的に行われるD論発表会=公聴会=ディフェンスだと、だいたい60〜90分だと思います。
・(ストレートで)26,7歳にもなって学生は…
▶︎周りは就職してしまって焦るかもしれませんが、Dで実力をつけてから就職するのもありだと思いますよ。
・いやいや、Dからじゃ就職できないよ
▶︎そんなことはありません。多くの企業がD卒生の採用を行っています。
・でもお金無いし
▶︎学振という制度や、RA等の制度が充実してきています。修士から就職した人と比べるとそこまでは稼げませんが、大学に通って研究してお金がそれなりに貰えます。
・生涯年収減るじゃん
▶︎修士に行かずに学部で就職した人と修士課程を比べたら学部で就職した方がおそらく上なので、それはそう、としか言えないですが、逆転の可能性もあると思いますよ。
・大学の先生にはなれるの?
▶︎企業の就活と同じく実力と運で左右されるので難しいですが、頑張ればなれると思います(個人の感想です)。
・どんな人がD進に向いてるの?
▶︎研究が好きなら是非。世界の人々の知にあなたの新しい発見を刻みましょう。
・D取ったら研究職しかないの?
▶︎そんなことないです。開発や営業やってる人だっています。研究に広く関係するものとして、ジャーナリストや作家、役人、出版社などに行った方でD持ちの方も多くいます。政治家にもDいますね。
・でも大変そう
▶︎たしかに大変かもしれませんが、Dを取ったという経験はあらゆることへの自信に繋がります。貴重な20代を、と思うかもしれませんが、貴重な20代に投資した、という考え方もできると思います。
・その他
(AC担当日まで、twitterで質問が来たら随時ここに記載します)
+ + 以下追記 + +
・Dで研究室を変える/研究テーマを変える場合に覚悟しておくべきことは?
▶︎論文をどう出していくか、指導教員とよくよく相談しましょう。M→Dと続けてきた学生は、修士時代の研究をそのままDの間に論文にし、Dの論文カウントに加えたりすることができますが、Dで研究テーマが変わった場合にはそれが通用しなくなります。論文が2本要るなら、少なくともD2の夏に1本投稿、D3の夏にもう1本と進めていかないと3年を超える恐れもあるでしょう。
・あと、修士の時に書いたプロシーディングってDで業績カウント出来たりするんですか!?
▶︎カウントできる場合があります。修士+博士後期の5年間で博士課程という考え方があるためです。ただし、扱いは大学・部局によりますので、指導教員にご相談を。
・学振取れない場合の収入源をどうするか
▶︎学振が取れれば月20万のお給料が貰えますが、競争率も高いので、取れなかった場合の話になります。
似たような助成をしている財団等に申請する、理研JRAや産総研RAといった制度に申請するなど、いくつか手はあります。また、大学でTA/RAとして学生を雇用して給料を出すということもやっていると思います。大学RAの場合、原資は指導教員の研究費だと思うので、そこも指導教員と相談してみてください。
・D進する人って大体半年くらい留学してるイメージがありますが、一般的?
▶︎半々くらいかなと思います。私は留学をしたことがないので、羨ましくも思いますが、必ずしも留学経験がないといけないわけではないです。ただし、経歴(就活等)としては留学はプラスに働くことの方が圧倒的に多いです。
・結婚しなくてDに耐えられるでしょうか
▶︎猫を飼いましょう🐱
・課程博士社会人博士と論文博士の違い(取得難易度や価値の違いなど)
▶︎論博の方が難易度は圧倒的に上です。確かなことは言えないですが、多くの部局で論文数で倍くらいの要件があると思います。
・課程博士社会人博士論文博士において,D論における研究の一貫性/大テーマとはどれくらいの広さが求められるor認められるのでしょうか?
▶︎基本的に課程博士や社会人博士の関係なく、D論は1つのテーマとして完結していないといけません。一貫したストーリーが描けることもDの要素だと思います。
・社D時に修士と同じ方向性のテーマでD論を書く場合,修士のときの査読付きジャーナル論文などは使えるのでしょうか,という点について人により意見が異なることがあるようですが指導教官の意見が支配的とみるのがよろしいでしょうか?
▶︎指導教員+副査の先生方、要するにD論審査員の意向がほぼ全てです。部局での慣例もあるかもしれません。事前に指導教員に聞いておくと良いです。
・一貫性の定義とそれの評価は同分野の現物の博士論文が参考になるのでしょうか?
▶︎例えばテキトウな例ですが、自然言語と音声情報とで機械学習を適用したD論とかあるかもしれないです。手法開発という側面でサーベイしてストーリー付けしてるとか、なんらかの一貫性があるかと思います。多くの大学はウェブでD論を公開しています(数年前にウェブ公開が義務付けられました)。
・恋人・配偶者が存在しないとして,そういう人を作ろうとするつまり恋愛をするというのは,逆に(特に男性は)キケンと伺ったことがありますが,どうでしょうか?
▶︎個人の感想の域を超えないのですが、恋人のために感情が揺れ動くことは研究の上でも良い効果があるかもしれません。もちろん、恋愛なんかにうつつを抜かしている暇があれば研究しろ、という意見もあるかもしれないですが、プライベートはプライベートで切り分けて考えられる方が、今後の人生に良い方向にはたらくと思います。
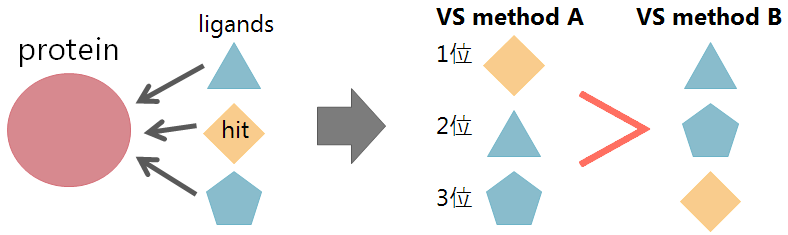
バーチャルスクリーニングで使う構造の賢い選び方?
この記事は今年読んだ一番好きな論文 Advent Calendar 2015の23日目の記事です.
今日紹介するのは,Journal of Chemical Information and Modelingという論文誌に掲載されたAn Inexpensive Method for Selecting Receptor Structures for Virtual Screeningという論文です.日本語で言うと,「バーチャルスクリーニングで使うタンパク質の構造を割と軽めの計算で選ぶ方法」というものです.
Huang Z, Wong CF. J Chem Inf Model. (in press), doi:10.1021/acs.jcim.5b00299
Publication Date (Web): December 14, 2015
http://pubs.acs.org/doi/10.1021/acs.jcim.5b00299
先日アクセプトされたばかりでまだ著者原稿版しか載っていませんが,僕らがやっている研究に近いというか,なんで思い付いてさっさと投稿しなかったんだというツッコミを自分に入れながら読んでいました.
1 バーチャルスクリーニングとは?
バーチャルスクリーニング (virtual screening) とは創薬分野で主に使われる単語で,計算機で薬の候補になりそうな化合物を選別 (screening) することを指します.薬の候補になりそうかどうかは,あるターゲットのタンパク質に対して活性が有るか無いか,という指標で図られます.この活性の有無を予測して選別することが目的です.
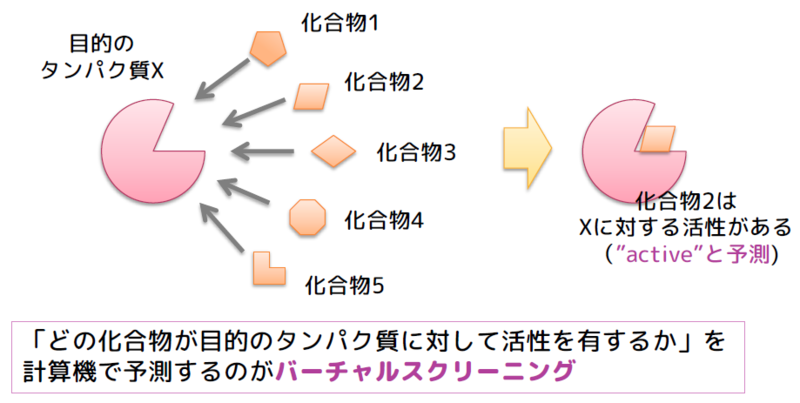
バーチャルスクリーニングには大きく2つの方法があり,化合物の形と既に分かっている活性の情報(教師情報)から未知の化合物に対する活性を予測するligand-based drug design (LBDD) と,ターゲットとなるタンパク質の立体構造情報を使ってドッキングシミュレーションなどの物理化学的な計算を用いるstructure-based drug design (SBDD) があります.それぞれ一長一短ありますが,今回紹介する論文は後者のSBDDのお話です.
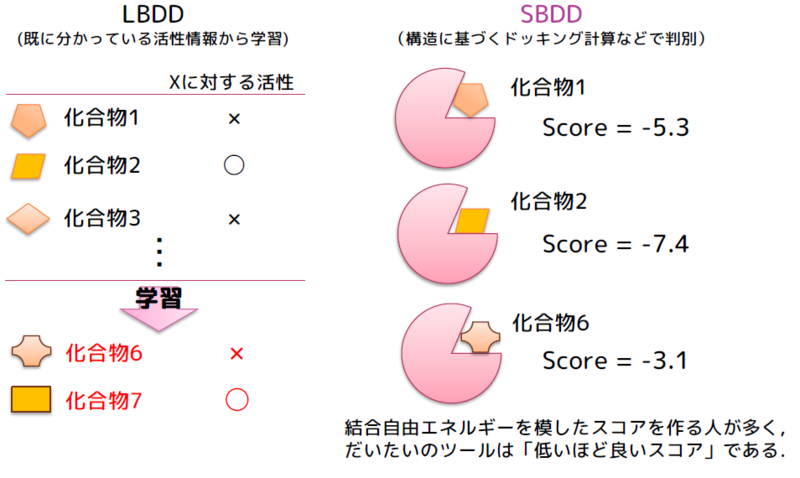
2 この論文はどういう問題を扱ってるの?
SBDDでは,ターゲットとするタンパク質の立体構造がとっても重要です.立体構造を決めた人はProtein Data Bank (PDB) というデータベースに登録していくのですが,同じタンパク質でもいろんな立体構造があるので,それらは個別にそれぞれ登録されています.構造屋さんはリゾチームが大好きなので,例えばリゾチームを見てみると700個くらい立体構造がPDBに登録されています(参考:http://d.hatena.ne.jp/tonets/20120730/1343655777).
そのため,「あるタンパク質Xを阻害する化合物を探したい!」と言っても,タンパク質Xの構造データはたくさんあるので,そのうちどれを使ってSBDDすればいいのか分かりません.化合物がはまりそうなポケットに何か既にはまっているもの(ホロ体といいます)だと,そうでないもの(アポ体)よりも良さそうですが,一概には言えません.どの構造がバーチャルスクリーニングに適しているかを選ばなければなりません.

上の図はイメージ図ですが,実際の構造もちょっとずつ違っています.CDK2タンパク質を例に見てみましょう.

ほとんど同じと思う人も多いかと思いますが,ちょっとずつ違っています.
3 1番単純な方法
さて,構造の選び方ですが,1番シンプルな方法はこんな感じです.
タンパク質Aについて既に活性がある化合物(active)と,活性がない化合物(inactive)または活性が多分ない化合物(decoy)を集めてきて,実際にタンパク質Aの構造A1, A2, ...とドッキングさせて「activeの評価が高く,inactive/decoyの評価が低くなる」ようなタンパク質の構造を選べば良い.
図にするとこんな感じです.化合物がスコアの良い順に並んでいると思って下さい.
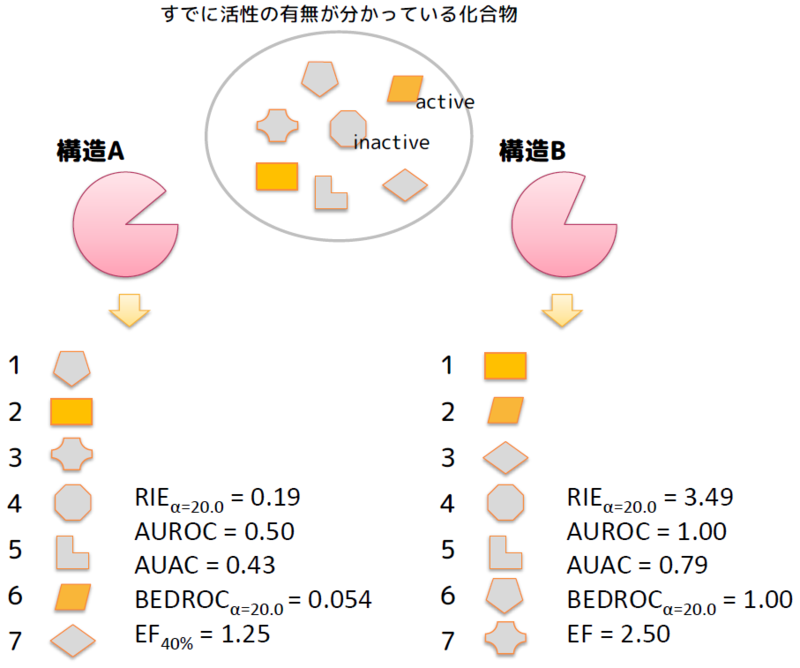
図中にでているRIE,AUROC,AUAC,BEDROC,EFはどれもランキングの良さを表す指標で,上位にactiveが来れば来るほど大きな値になります.詳細はhttp://d.hatena.ne.jp/tonets/20140604/1401856579とか見て下さい.
いろいろ指標を出しましたが,まぁ人の目で見ても「構造B」が良さそうというのが分かりますね.
4 1番単純な方法の問題点
この方法は1番単純でかつ確実な方法なのですが,計算が大変という問題もあります.図ではactiveが2個でinactive (decoy) が5個ですが,実際にはactiveが数十個,inactive (decoy) は数千個というレベルで計算させることが多いです.単純に数が多いので大変,ということですね.
5 この論文が提案したこと
この論文では,「activeとinactiveを全部ドッキングするのは大変だから,activeだけの結果からなんとか判断しよう」としました.5つの指標を提案していて,そのうちScreening Performance Indexと名付けた5番目の指標が1番良かったと言っています.SPIの式をそのまま引用します.
はactiveの数,
はドッキングがちゃんとできたactiveの数ですがほとんど
と同じです.
は
を全ての候補の構造で足し合わせたもので,構造が
個あって
個のactive化合物が全てドッキングできたとすると
となります.
式で見るとちょっと複雑そうですが,要するにactiveだけドッキングした結果の全体平均スコアよりも高いスコアになったactiveをたくさん得た構造が勝ち,ということです.図にするとこんな感じです.

この図では,平均が-8.7で,構造Aは長方形の化合物だけ,構造Bは2つとも平均より良いスコアなので,構造Bの方がSBDDに適していると言うことができます.
6 SPI (Screening Performance Index) を使った結果
さて,本当にこのSPIという値でバーチャルスクリーニングに適した構造を選ぶことができるのでしょうか.詳細は割愛しますが,この論文では8種類のタンパク質に対して,それぞれ10〜30個くらいの構造を用意して,activeだけを使ってSPI値で選んだ構造が,実際にactiveとinactiveの両方を使って計算したBEDROC/RIE/AUAC/1%EF/10%EFとどのくらい相関するかを示しています.結果的にはSPIとBEDROCが平均して0.87ほどの相関係数を持つことが示されました.つまり,activeだけで選んだ構造は,実際にactiveとinactiveの両方で検証しても識別能が高かったということになります.
興味深いのは,結合部位の体積やタンパク質構造の解像度,アポ体/ホロ体の区別とはあまり関係がなさそうだったということです.このあたりは特徴付けが難しいのですが,構造の特徴そのものから識別能が分かるようになると,activeとのドッキングすら要らなくなるので,More Inexpensiveな方法で構造を選ぶことができるようになります.この論文の将来展望といったところでしょうか.
7 あとがき
細かい方法論の論文を紹介してしまいましたが,意外に誰もやっていなかった話(もしくはみんな暗黙のうちにやっていたかもしれない方法)をうまく論文化したなぁという印象です.ちなみにこのJournal of Chemical Information and Modelingという論文誌は,JACSで有名なACSが刊行する雑誌で,ケモインフォマティクスを中心に,分子シミュレーションやバイオインフォマティクスの方法論も数多く載せています.バーチャルスクリーニングとか言い出す人はまず読んでいる雑誌なので,もしこういった分野に興味がありましたら論文を眺めてみると良いかもしれません.
博士課程一問一答(tonetsの場合)
以下,国立情報系で博士を取ったポスドクについて.
http://anond.hatelabo.jp/20141201200815 から一部拝借,補足的な感じで.
Q1. 博士課程ってなんですか
A1. 博士後期課程の略。学部4年、修士課程2年過ごしたあとに、さらに3年間(医薬系は4年間)大学に在籍して研究を行う。
Q2. なんで博士課程に進学したんですか
A2. 修士の後にふつうの企業で働くというイメージが湧かなかったから,と,研究を続けたかったから.
高校(高専)時代から漠然とD進する意思はあったしその時から親の理解もあった(ここ重要).
Q3. 博士取ったあとは?
A3. 無職(仮)です.(あとで詳細を)
Q4. 在学時の金銭問題に関して一言
A4. 博士課程で重要なのは,言うまでもなく「衣食住(生活費)」と「学費」です.一部の裕福な人以外は多分みんなカツカツだと思います.
学費は大学の授業料免除制度を積極的に活用しましょう.ない場合は諦めましょう.(TRA系のサポートがある大学も多いです.)
生活費は僕の場合は学振DC1です.D1〜D3の3年間,月20万円もらっていました(給与扱いです).
最近はリーディング大学院の奨励金(給与扱い)や,ラボの研究費でRAとか,いろいろ工面する手段があります.なかったらバイトですね.
Q5. 学振とは?
A5. 日本学術振興会特別研究員.学生向けにDC1,DC2の2つ,ポスドク向けにPD, SPD, RPD, 海外学振の4つがある.
DC1はM2(医薬系はD1でも可)で申請,通ればD1から3年間20万円+研究費が貰える制度.
DC2はD1, D2の時に申請して,通れば翌年から2年間20万円+研究費が貰える制度.
PDはD3の時に申請して,通れば翌年から3年間36.2万円+研究費が貰える制度.SPD/RPD/海外学振は割愛します.
詳しくは,こちらのスライドを.[学振]でググると割と上位に出てきます. http://www.slideshare.net/tonets/10tips-32604093
僕の場合は,M2のときにDC1を申請して,面接を経て採用.D3のときにPDを申請して面接免除で採用,でした.
学振を貰っていると大学の授業料免除ができないという噂もあるので,簡単なアンケート調査もやったことがあります. http://goo.gl/GZVzmv
このあたりは口コミ情報でしか得られない話なので,行きたい大学・部局の先輩によく聞きましょう.
Q6. 博士課程はどんな感じでしたか?
A6. 気ままに研究してたと思います.今とあんまり変わりません.
自分の分野がいわゆる学際領域だったので,関連する分野のいろんな学会に足を伸ばしました.
国内だと,日本バイオインフォマティクス学会,日本蛋白質科学会,情報処理学会,生物物理学会を中心に,情報計算化学生物学会,ハイパフォーマンスコンピューティングと計算科学シンポジウム,NVIDIA GTC Japanなど,国外だとISMB, InCoB, APBC, ACM-BCB, PRIB, GIW, GLBIOなどです.とにかく生物分野の知識が圧倒的に無かったので,国内の若手の会と呼ばれる学生・若手研究者の集まりにもたくさん参加して時に主催側になったりしながら勉強しました(生命情報科学若手の会,生物物理若手の会,生化学若い研究者の会,タンパク質構造理論勉強会,W8F5seminar).特に若手の会での人との繋がりは研究を進める上でも重要な役割を果たしていたと思います.
Q7. で,今はなにしてるの?
A7. 上の学振でPDとして採用されていわゆるポスドク(研究員)をやっています.
なぜ無職(仮)と書いたかというと,雇用契約が無いからです.大学からも学振からも雇われていません.
例えば大学職員・学生が安く買えるソフトウェアとか買えません.うちの大学のスパコンに入ってる商用ソフトウェアも使えません.
年金も1階だけです.健保も自分で払います.大学での健康診断とかももちろん受けられません(この辺は機関によるかな).
不動産を買いたくてもローンなんて絶対組めません.
それでも良ければ学振PDをおすすめします.何より自由が手に入ります.
Q8. 楽しいこと3つ
A8.
(1) 自分の時間を好きに使える.時間に縛られない.
(2) 好きな研究ができる.
(3) 世界を変えられる.
Q9. つらいこと3つ
A9.
(1) 世界を変えられない(変えるほどの研究成果が生まれない)
(2) 英語が酷い
(3) 「大学に残ってるの?教授?」
Q10. 現時点で後悔していること
A10.
(1) 学生時代に留学しなかったこと
(2) 学生時代に雀荘のメンバー(アルバイト)をしなかったこと
(3) 学生時代に麻雀をやめなかったこと
Q11. やってよかったこと
(1) ダメ元でチャレンジしたこと.博士の終わり頃に取った育志賞もダメ元でした.
(2) たくさん発表したこと.発表機会を与えてくれたボスにも感謝です.
(3) 結婚
以上です.聞きたいことがあればまた書きます.
バーチャルスクリーニングの指標のテスト その2
昨日の記事の続きです.なんの話かということは昨日の記事を参照のこと.
(バーチャルスクリーニングの指標のテスト)
今日はさらに指標を4つ紹介する.AU-ROC,AUAC,BEDROC,EFの4つ.
AU-ROC (Area Under the Receiver Operating Characteristic Curve)
みんな大好きROC曲線のAUC.
バーチャルスクリーニング(early recognition problem)でのROC曲線は,横軸にヒットじゃない化合物の数,縦軸にヒット化合物の数を引いてプロットする.
例えば「リガンドの数10,ヒットの数5,ヒットの順位は1,3,4,6,9だった」ときはこんな感じ.

で,この曲線の下の面積がAU-ROC (AUCと呼ばれることの方が多いが,) である.
で,このAU-ROCはまぁ一応積分計算なのであるが,離散問題なので簡単に計算できる.
はヒットの数,
は(
個目の)ヒットの順位,
はテストした化合物の数である.
有名でよく使われる指標だが,バーチャルスクリーニングの指標としてはあまり良くない,と言われている*1.
AUAC (Area Under the Accumulation Curve)
あんまり有名じゃないしあんまり使われてない指標.ROC曲線は横軸にヒットじゃない化合物の数をとっていたが,Accumulation曲線は横軸に(全ての)化合物の数をとる.
例えばさっきの例,「リガンドの数10,ヒットの数5,ヒットの順位は1,3,4,6,9だった」はこんな感じ.
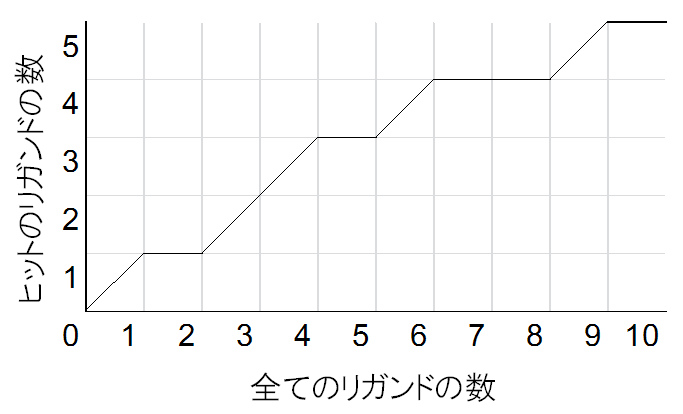
Accumulation曲線のときは線は斜めに引く.この曲線下面積AUACも,もちろん簡単に計算できる.
はヒットの数,
は(
個目の)ヒットの順位,
はテストした化合物の数である.
やっぱりバーチャルスクリーニングの指標としてはあまり良くない,と言われている.
なお,だと AU-ROC≒AUAC となる.そういうわけでAU-ROCを使ってればOK,なのだ.
BEDROC (Bolzmann-Enhanced Discrimination ROC)
昨日紹介したRIEを0-1に正規化した感じの値.
はRIEで使っているパラメータである.
BEDROCは結構良い指標らしい.最近は良く使われている.
EF (Enrichment Factor)
この界隈ではかなり有名.結構良い指標と言われている.
はEFのパラメータで,上位
%を見て計算する,という意味になる.
#ちなみにmimeTeX記法でなぜか"#"が入力できなかった."\#"でも駄目らしい.countは#と同じ意味.
例えば「200個のhitが10000個のligandの中にある.15%(30個)のhitが上位5%(500位以内)に存在した.」というときのEFは,
,
なので,
となる.
EFの最大値は,ランダムのときはほぼ1で正確には
となる.
4つの指標をテスト
前回のプログラムをちょっと変更して今回紹介したパラメータを計算できるようにした.
#!/usr/bin/python import sys from math import * if len(sys.argv) != 3: print 'Usage: %s [N] [list of hits (ex: 2,3,5)]' % sys.argv[0] quit() N = float(sys.argv[1]) hits = sys.argv[2].split(",") n = float(len(hits)) ec = 0 # EF count arp = 0.0 rie = 0.0 alpha = 8.0 # RIE paramater chi = 0.2 # EF parameter for ri in range(1, int(N)+1): if str(ri) in hits: arp += ri rie += exp(-alpha*ri/N) if ri <= chi*N: ec += 1 ri += 1 arp = arp / n rie = rie / ((n/N)*((1-exp(-alpha))/(exp(alpha/N)-1))) auroc = 1 - arp/(N-n) + (n+1)/(2*(N-n)) auac = 1 - arp/N bedroc = rie * (n/N) * sinh(alpha/2)/(cosh(alpha/2)-cosh(alpha/2-alpha*n/N)) + 1/(1-exp(alpha*(N-n)/N)) ef = float(ec)/(chi*n) print "ARP =", arp print "RIE =", rie print "AU-ROC =", auroc print "AUAC =", auac print "BEDROC =", bedroc print "EF =", ef
今回はリガンドを10個,ヒットが4個で試してみる.
Aのヒット順位は(1, 2, 5, 9),Bのヒット順位は(2, 3, 5, 6)だったとしよう.で計算する.
$ python virscr2.py 10 1,2,5,9 ARP = 4.25 RIE = 2.05435170462 AU-ROC = 0.708333333333 AUAC = 0.575 BEDROC = 0.855180830287 EF = 2.5
$ python virscr2.py 10 2,3,5,6 ARP = 4.0 RIE = 0.978186814512 AU-ROC = 0.75 AUAC = 0.6 BEDROC = 0.402850472687 EF = 1.25
というわけで,
である.指標によって変わるが,ARPやAU-ROC, AUACは少数でも後ろの方の順位のヒットがあると引きずられやすいため,Bの方が良くなっている,と言えるだろうか.逆にRIE, BEDROC, EFは,パラメータを使って上位にヒットがあることを考慮して値を出しているためAの方が良いという結果になると言えると思う.
おとなしくEF(かBEDROCあたり,もしくはもっと新しいもの)を使いましょうということだ.
*1:Truchon J-F, Bayly CI. Evaluating virtual screening methods: good and bad metrics for the "early recognition" problem. J Chem Inf Model, 47: 488-508, 2007. とか
バーチャルスクリーニングの指標のテスト
久しぶりの記事.久しぶりすぎたのでテンプレも変えた.
最近バーチャルスクリーニング(virtual screening)という話を勉強中である.
簡単に言うと,薬(でもなんでも)のモトになるligand(とか)を計算機で選別しましょう,というもの.
話すと長くなるので詳細は割愛するが,例えばprotein-ligand dockingなんかで有望そうな化合物(ligand)を選別したいのだ.この有望そうな化合物はよく「ヒット化合物」とか言われる.
選別する手法はものっすごくたくさんあるのでこれも割愛するが,どんな手法が良いのかの基準というものがないと手法の良し悪しが測れない.
手法の良し悪しは「early recognition problem」として評価することが多いようだ.early recognition problemとは,端的に言えば「上の方の順位にヒットがいたら優秀」というわけだ.
イメージはこんな感じ.
この例だとぱっと見で手法Aが優秀だとわかるが,実際はもっとごたごたしている.もっとたくさんの化合物を試すし,ヒットは複数ある場合も多い.
というわけなので,定量的に評価する指標が存在する.今日は2つ紹介する.ARPとRIEである.
ARP (Average Rank of Positives)
なんか大層な名前が付いているが,要はヒットの順位の平均である.
はヒットの数,
は(
個目の)ヒットの順位である.
定義から分かる話だが「小さい方が良い」値である.簡単に求められるし直感的だが,テストした化合物の数やヒットの数などでどのくらいの値が良いという話が変わってくるのであまり実用的ではない.
RIE (Robust Initial Enhancement)
こちらはもうちょっとマシな指標であるが式はごたごたしている.
はヒットの数,
は(
個目の)ヒットの順位なのはARPと同じ.
はテストした化合物の数,
は謎パラメータである.一応Enrichment Factorという別の指標の
の逆数と似たような意味,という意味合いのパラメータではあるが,今回は厳密な意味は割愛する.だいたい
とかが選ばれる.8.0だと「上位20%ぐらいに入ってて欲しいなぁ」という話になる(はず).
RIEは「大きいほど良い」値である.ランダムだと1になることが証明されている.
(というか元々は分母は分子の値をモンテカルロで1000回とかランダム計算してそのアンサンブル平均にしてね,という話だったのを,Truchon J-F and Bayly CI, J Chem Inf Model, 47:488-508, 2007 でちゃんと閉じた形で計算できるようになった,というわけ.)
さて,この2つの指標をテストするpythonプログラムをサクッと書いた.
#!/usr/bin/python import sys import math if len(sys.argv) != 3: print 'Usage: %s [N] [list of hits (ex: 2,3,5)]' % sys.argv[0] quit() N = int(sys.argv[1]) hits = sys.argv[2].split(",") n = len(hits) ri = 1 arp = 0.0 rie = 0.0 alpha = 8.0 # RIE paramater for i in range(1, N+1): if str(i) in hits: arp += ri rie += math.exp(-alpha*ri/float(N)) ri += 1 arp = arp / float(n) rie = rie / ((float(n)/float(N))*((1-math.exp(-alpha))/(math.exp(alpha/(float(N)))-1))) print "ARP =", arp print "RIE =", rie
最初の引数が,次の引数がカンマ区切りのヒットの順位である(スペースを入れると動かない).
$ python virscr.py 10 1,2,4,6 ARP = 3.25 RIE = 2.14608273845
のように使う.
例えば

の結果からAとBのどちらが良いかというのを見てみる.(見た瞬間にAの方が優秀だということが分かるが,値で確認してみよう.)
$ python virscr.py 4 1,3 ARP = 2.0 RIE = 1.76159415596
$ python virscr.py 4 2,3 ARP = 2.5 RIE = 0.265802228834
というわけで,ちゃんとAの方が「ARPは小さく」,「RIEは大きく」なっていることが分かる.
PyMOLのprotscaleに新しいカラーパターンを追加する
PyMOL1.3のrToolsというプラグイン集のcolor_protscaleプラグインで疎水性残基の色分けとかしてたけど,白-青とか赤-青みたいなよく見るやつしかなく,今回灰-白というカラーパターンが必要になったので作った.
まず
C:\Program Files (x86)\PyMOL\PyMOL\modules\pmg_tk\startup\color_protscale.py
を開く.
def get_color_mapのサブルーチンに
if name=='gray':
red = (1.0-assign_map[k])*0.6+0.4
green = (1.0-assign_map[k])*0.6+0.4
blue = (1.0-assign_map[k])*0.6+0.4
color_map[k] = [red,green,blue]
message = 'gray=1.0 white=0.0'
を追加.
def __init__(self):に
self.menuBar.addmenuitem('Color schemas', 'command',
'gray',
label='gray - white',
command = lambda : set_colorschema('gray'))
を追加.
これで「灰色〜白色」のカラーパターンができる.
PDBとUniProtのエントリ数あれこれ
PDB (Protein Data Bank) は実験的に決定されたタンパク質(など)の構造情報を提供するデータベースのこと,UniProt (Universal Protein resource) とはタンパク質の配列情報と機能アノテーションを提供するデータベースのことである.最近PDBの構造情報について,「ヒトのタンパクって何個くらいあるの?」とか「世のタンパク質のどのくらいの割合で構造がとれてるの?」とか良く聞かれるので,ちょっと調べてみた.
ちなみにPDBの統計情報については,ウェブからある程度は見れるようになっている.
- Protein Data Bank / http://www.rcsb.org/pdb
に行って,右上の方にある『PDB Statistics』をクリックすればいろいろ情報が取れるし,簡単な条件指定でエントリ数をかぞえるなら
- 『there are XXXXX Structures』
の数字(XXXXX)をクリックして,Query Refinementsの条件のところをポチポチ押していけばいい.
こんな感じの操作でヒトのタンパク質の数ぐらいならすぐ分かる.2012/7/24 ver. では,構造は全部で83,266個,うちタンパク質だけ(DNA・RNAやそのタンパク質との複合体などを除いたもの)の数は77,057個,さらにそのうちヒトのタンパク質(Homo sapiens only)は18,774個である.個人的には,意外と多い…という印象だ.
さて,世のタンパク質(UniProt)の数に対してPDBがどれだけの割合でとれているのかを調べるには,実際の構造情報を使って調べる必要がある.とりあえず,PDBをミラーリングダウンロードした.
だいたい一晩で落とせると思う.83,266個の.pdbフラットファイルだけだと15GB,XMLやmmCIF形式,またbiological unitなど全部含めて146GBであった.
この.pdbファイルに対して,Chainの情報を抽出したりChainに対応するUniProtIDの抽出をしたりを,テキトウなスクリプトで実行した..pdbはあんまり綺麗でないのでパースに失敗したりしてるのもあって概算でしか出せてないが,大体以下のような数字が分かった.
- UniProtIDを持つPDB Chainの数:195,656
- UniProtIDが付加されたChainを持つPDB数:77,193
- PDB中のユニークなUniProtID数:27,859
- PDB 全エントリ数:83,266
- UniProt 全エントリ数:536,789
というわけで,UniProtのエントリに対して,27,859/536,789≒5.2% しか構造が対応していないということが分かった.感覚で10%はあるものだと思っていたので,結構意外であった.
そんなわけで,UniProtIDが紐付いたChainが195,656個に対して,ユニークなUniProtID数が27,859ということは,結構な数の重複があるということである.そのヒストグラムをプロットしてみた.

これは横軸に「あるUniProtIDに対応しているPDB Chainの数」,縦軸に「UniProtIDの数」を取ったものである.右は左のグラフを両対数プロットしたものである.両対数で直線に乗るような感じ,である.
なお,PDB Chainの数が多いUniProtIDベスト5は,
- 698個 P01308 Insulin
- 696個 P00734 Prothrombin
- 646個 P00720 Lysozyme
- 542個 P19491 Glutamate receptor 2
- 509個 P61769 Beta-2-microglobulin
であった.パースがうまくいってないものが混じっていて概算でしかない(大事なことなので二回言う)が,500個超えとか,なかなか激しいなと思う次第である.